Ensinando Neuroanatomia Com o Computador
Luiz Gustavo Bizarro Mirisola e Francesco LangoneDez anos atrás, a Associação Americana de Escolas Médicas declarou ser importante que as escolas de medicina fizessem tudo para promover o uso efetivo da informática entre seus alunos e docentes1. De fato, durante esse período os recursos da informática deixaram de ser uma novidade para se tornarem uma ferramenta indispensável e rotineira em várias disciplinas das escolas médicas dos Estados Unidos2-4. Estudos recentes constataram que o conhecimento de informática dos alunos ingressantes nos cursos médicos tende a aumentar em relação aos últimos anos3,5,6.
Por outro lado, esses trabalhos mostram também que existe ainda um contingente significativo de alunos ingressantes sem qualquer familiaridade com computadores, um fenômeno que parece ser ainda mais significativo no Brasil. Em nosso meio, apenas 28% dos ingressantes da Unicamp no curso de Medicina e 7% no curso de Enfermagem eram usuários de computadores em 199410.
Diante deste quadro consideramos importante, e mesmo necessário, estimular desde o primeiro ano do curso o emprego de estratégias de ensino que lancem mão das alternativas oferecidas pela informática esperando otimizar significativamente o tempo de aprendizado e a fixação dos conceitos apreendidos, considerando ainda que só o contato dos estudantes com os computadores já é per se benéfico à sua formação.
Dentre as disciplinas do primeiro ano dos cursos da área da saúde, a Anatomia é uma das mais apropriadas para a introdução do estudante à informática9,11,12.
Até o presente, na maioria das escolas médicas do Brasil, a Anatomia vem sendo ensinada através de aulas expositivas, gravuras, diapositivos, atlas, livros textos, que nem sempre podem ficar à disposição dos estudantes para estudo ou revisão, ou cortes histológicos e peças dissecadas, que são de difícil compreensão para a maioria dos alunos ingressantes. Este fato, em particular, exige a presença constante de, em media, 1 docente para cada 15 alunos durante a aulas práticas para que haja um grau razoável de aproveitamento. Essa proporção, entretanto, dificilmente é atingida (na Unicamp é 1 docente para cada 25 alunos) e está piorando. Na Neuroanatomia, particularmente, esse dado é muito importante, pois, mais do que em qualquer outro sistema, o estudo da morfologia não pode ser dissociado da função e, no caso do curso médico, se faz necessária muitas vezes a abordagem de aspectos patológicos e clínicos para a sua melhor compreensão, obrigando o estudante a trabalhar conceitos realmente novos e que exigem um esforço maior no seu aprendizado.
O presente artigo exemplifica este tipo de aplicação através de um software desenvolvido pelos autores, que contém ilustrações gráficas e informações teóricas sobre o encéfalo humano, e que permite, de modo interativo, ao estudante trabalhar autonomamente os conceitos básicos da Neuroanatomia Funcional. Explicamos em linguagem simples alguns dos detalhes técnicos, que podem servir como uma orientação para os que quiserem realizar projetos semelhantes de atlas digitais em qualquer domínio da biologia e da medicina.
Aquisição das Imagens
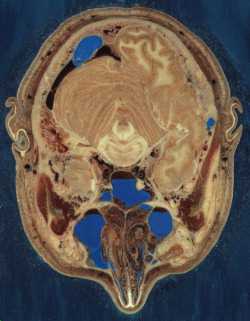
O primeiro passo na construção de atlas anatômico ou patológico consiste na obtenção das preparações a serem fotografadas digitalmente. Em nosso caso, tínhamos a intenção de fazer um atlas seccional do encéfalo. Para isso, usamos material anatômico disponibilizado pelo Departamento de Anatomia do Instituto de Biologia da Unicamp. Foram utilizados dois cadáveres humanos, do sexo masculino, fixados em formol. De cada cabeça, congelada separadamente, foram feitos cortes seriados com 1,0 cm de espessura segundo os planos axial, sagital e frontal, de acordo com os padrões seguidos na tomografia computadorizada.A aquisição das imagens foi feita utilizando-se um scanner colorido Microtek ScanMaker II, e o software Adobe Photoshop 3.0. A digitalização das imagens foi feita a 150-300 dpi (pontos por polegada) e depois reduzida para a resolução da tela (72 dpi), em formato JPG (escolhido por ser o formato que apresenta maior compressão dos arquivos, resultando numa economia de espaço de memória e tempo de acesso a disco). Estas imagens possuem tamanhos em torno de 50 a 150 Kb, embora quando estas são descomprimidas para apresentação na tela, cada uma delas ocupa de 2 a 5 Mb de memória. As imagens digitalizadas até o presente momento correspondem a todos os cortes das três cabeças inteiras, correspondendo a 10 cortes no sentido frontal, 12 cortes no sentido axial e 12 cortes no sentido sagital. As imagens foram processadas pelo PhotoShop, ajustando-se o seu contraste, brilho e balanço de cores, de forma a evidenciar os bordos e a resolução de estruturas anatômicas, facilitando a visualização do usuário.
Além disso, para cada imagem existe um esquema correspondente da mesma, utilizado para identificar e rotular as estruturas anatômicas nele constantes, e que foi obtido a partir do corte do cérebro com o auxilio do Photoshop (vide figura). Obtivemos uma imagem em preto e branco onde estão presentes apenas os contornos das estruturas contidas na mesma (ou seja, os pontos onde há uma variação brusca de cor sobre a imagem). A seguir, na posição de cada estrutura se escreveu o seu numero correspondente em vermelho, para servir como referência ao usuário.
Desenvolvimento do Software
O passo seguinte foi desenvolver um software para Windows, capaz de visualizar as imagens e esquemas, e servir como ferramenta de navegação e de estudo. Para esse desenvolvimento, escolhemos o Delphi 2.0 (da empresa Borldand), por apresentar melhores facilidades para se implementar uma aplicação Windows com suporte a bancos de dados. O Delphi possui uma biblioteca de componentes com toda sorte de botões, janelas, caixas de texto, etc.., presentes numa janela Windows 95, além de componentes de acesso a banco de dados. Permite ainda o emprego de componentes específicos, obtidos via Internet, ou que desenvolvamos nossos próprios componentes. Utilizamos também a linguagem Borland Object Pascal, que permite a utilização de classes e objetos.A base de dados foi desenvolvida usando o software de geranciamento Microsoft Acess 95. Ele acessa duas tabelas. A primeira referencia as imagens. Cada registro contém o nome do arquivo da imagem, e uma descrição, bem como um esquema associado (opcional). A segunda tabela contém as estruturas anatômicas, seus nomes, coordenadas e descrição, bem como os nomes das imagens onde ocorrem. As coordenadas descrevem figuras geométricas que se aproximam ao contorno da estrutura, como retângulos, círculos e polígonos fechados. Esse esquema facilita a definição de regiões em uma imagem numa página HTML, usada em browsers da Web. Assim, caso futuramente se deseje utilizar as mesmas imagens e estruturas numa página HTML o trabalho será muito facilitado.
Com exceção da tabela de imagens que, uma vez aberta, assim permanece, permitindo que o usuário navegue pelos seus registros, todas as consultas e alterações na base de dados são feitas usando a linguagem SQL (Standard Query Language), uma linguagem de acesso a banco de dados que é suportada pelo Delphi.
Funcionamento do Software
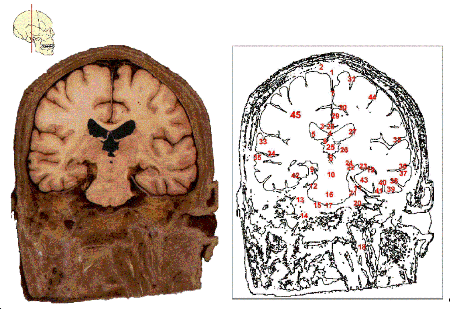
Exemplo de tela do sistema mostra corte anatômico e esquema com legendas
O software possui uma janela principal e várias janelas auxiliares, que vão se abrindo à medida que o usuário solicita funções específicas, como um teste de seus conhecimentos, ou buscas por palavras-chave. A janela principal apresenta dois visualizadores de imagens, um para a imagem do corte anatômico e outro para o esquema de identificação das estruturas. Uma vez carregadas as imagens, podemos utilizar um controle na barra de ferramentas da janela para aumentar ou diminuir o zoom, de 1/16 até 16 vezes o tamanho original da foto.
Há também um componente navegador, com botões para se avançar e retroceder um registro na base de dados, para se excluir um registro (cada registro, lembramos, corresponde a uma imagem com o seu esquema), etc. Na barra de status, na parte inferior da janela, são mostrados a coordenada corrente do cursor do mouse sobre qualquer uma das duas imagens mostradas, e na parte esquerda a "dica", ou seja, um pequeno help sobre o botão ou opção de menu, ou ainda, caso esta opção esteja selecionada, o nome da estrutura que esteja sob o cursor do mouse no momento.
O menu apresenta opções para se abrir uma tabela, para acrescentar uma imagem a uma tabela aberta, e para se mudar os arquivos de imagem do registro atual. Alem disso, o menu apresenta opções para o usuário escolher o modo de operação:
Modo normal, em que ao se clicar sobre uma estrutura é apresentado um texto descritivo sobre a mesma (campo de descrição da tabela de estruturas);
Modo de entrada de novas estruturas, onde aparecem três novos botões na barra de ferramentas, que permitem selecionar entre retângulos, círculos e polígonos. Quando ele terminar de definir a nova estrutura ser-lhe-á perguntado o nome assumido por essa estrutura.
Modo de edição de estruturas: primeiramente serão desenhadas sobre a imagem os contornos de todas as estruturas presentes sobre aquela imagem, e serão mostrados na barra de ferramentas quatro botões, indicando se o usuário quer apagar estruturas, editá-las, ou mudar o nome delas. O menu apresenta também itens para se abrir as outras janelas do programa: os itens Testes; Sobre; e Pesquisar.
O software também permite realizar testes de conhecimento de anatomia seccional do cérebro pelo usuário. Ele apresenta uma estrutura anatômica e pede a sua identificação ou vice-versa, e contabiliza o número de acertos e erros que o usuário têm até o momento, apresentando os números na parte superior da tela juntamente com uma barra de progresso que indica a porcentagem de acerto.
Outra tela do programa permite realizar uma consulta por palavras-chave à tabela de estruturas, mostrando como resultado os nomes da imagens correspondentes. Por exemplo, se existem várias imagens com uma estrutura chamada "Tálamo", e deseja-se que todas elas tenham o mesmo texto descritivo, o usuário pode entrar "Tálamo", ou "Tála".
Finalmente, implementamos também um Help on-line (janelas de ajuda) para o programa, havendo um tópico de help para cada janela e para item (Arquivo, Editar, Imagem, Janelas, Sobre) do menu principal do programa.
Conclusões
O programa aqui descrito atende aos requisitos de um software útil ao ensino de disciplinas básicas de morfologia (anatomia, histologia, patologia, etc.), permitindo armazenar as informações mais relevantes sobre os cortes anatômicos e as estruturas correspondentes para o seu estudo. Além disto permite imprimir e salvar as imagens ou os textos, que poderão ser empregados na elaboração de slides, atlas e material de estudo personalizado, etc.Consideramos ainda que o programa pode ser utilizado em quaisquer outras áreas do conhecimento que necessitem do armazenamento de informações sobre regiões em imagens, bastando para isso se conseguirem as imagens e se delimitarem as estruturas desejadas. O formato em que as informações são armazenadas permite que as imagens e as estruturas sejam utilizadas como base para a elaboração de novos projetos no futuro.
Os recursos do programa permitem que ele
seja de fácil operação, pois se destina a estudantes
dos primeiros anos das áreas biológicas que nem sempre têm
familiaridade com computadores. Interessados em adquirí-lo poderão
contatar o Centro de Documentação em Informática Biomédica
do Núcleo de Informática Biomédica da Universidade
Estadual de Campinas (Caixa Postal 6005, Campinas, SP 13081-970, Email:
nuri@nib.unicamp.br, Fax: (019)
788-7130.
Recursos de Ensino de Anatomia na InternetProjeto Ser Humano Visível. Site BrasileiroAnatomia do Corpo Humano e Tornozelo Interativo Demonstração de Anatomia Correlativa e Atlas de Ressonância Magnética de Anatomia Musculoesquelética, Slice of Life (Fatia da Vida), Videodisco interativo. A.D.A.M.,
Animated Dissection of Human Anatomy, um software e CD-ROM para
ensino de anatomia e cirurgia
Curso de Anatomia Humana, Emory University, Atlanta, EUA. Slides de Estudo de Neuroanatomia, Tulane Medical School, EUA. The Digital Anatomist , um programa de ensino com imagens 3D. Dissecções Cadavéricas em Anatomia Neurocirúrgica Beyond Vesalius, um curso de Anatomia Humana. |
Referências Bibliográficas
- Association of American Medical Colleges (1984): Physicians for the 21st century (Report of the Panel on the General Professional Education). Journal of Medical Education 59, 1-208.
- Association of American Medical Colleges. (1986): AAMC 1986-1987 Curriculum Directory.
- Piemme,TE (1988): Computer-Assisted Learning and Evaluation in Medicine. JAMA 260(3), 367-372.
- Klar,R; Bayer,U (1990): Computer-Assisted Teaching and Learning in Medicine. International Journal of Biomedicine and Computing 26, 7-27.
- Saltz,CC; Saltz,J; Rabkin,MR (1985): Perceptions and knowledge of medical students regarding computer aplications in medicine. Journal of Medical Education 60, 726-728.
- Kidd,MR; Connoley,GL; Cesnik,B; McPhee,W (1993): What do medical students know about computers? The Medical Journal of Australia 158, 283-284.
- O vestibular da UNICAMP em dados (1994). Comissão Permanente para os Vestibulares -Pró-reitoria de Graduação, UNICAMP.
- Vestibular Nacional 94 - Estatística do Questionário dos Alunos Matriculados por Curso (1994). Comissão Permanente para os Vestibulares - Pró-reitoria de Graduação, UNICAMP
- Guy,JF; Frisby,AJ (1992): Using interactive videodiscs to teach gross anatomy to undergraduates at the Ohio State University. Academic Medicine 67(2), 132-133.
- Kidd,MR; Cesnik,B; Connoley,G; Carson,NE (1992): Computer-assisted learning in medical education. The Medical Journal of Australia 156, 780-782.
- Lipscomb,K; Kittrel,A (1991): Software gets under your skin. Physicians Comput 9:14-16.
- Schubert, R.; Höhme, KH; Pommert,A; Riemer,M; Schiemann,T; Tiede,U; Lierse,W (1994): A new method for practicing exploration, dissection, and simulation with a complete computerized three-dimensional model for brain and skull. Acta Anatomica 150, 69-74.
Francesco Langone é professor do Instituto de Biologia
e coordenador-associado do Núcleo de Informática Biomédica
da Unicamp. Email: langone@turing.unicamp.br

